Este es un hilo relativamente antiguo, pero recientemente encontré este problema en mi trabajo y tropecé con esta discusión. La pregunta ha sido respondida, pero siento que el peligro de normalizar las filas cuando no es la unidad de análisis (ver respuesta de @DJohnson arriba) no ha sido abordado.
El punto principal es que normalizar las filas puede ser perjudicial para cualquier análisis posterior, como el de vecino más cercano o k-means. Para simplificar, mantendré la respuesta específica de centrar las filas en la media.
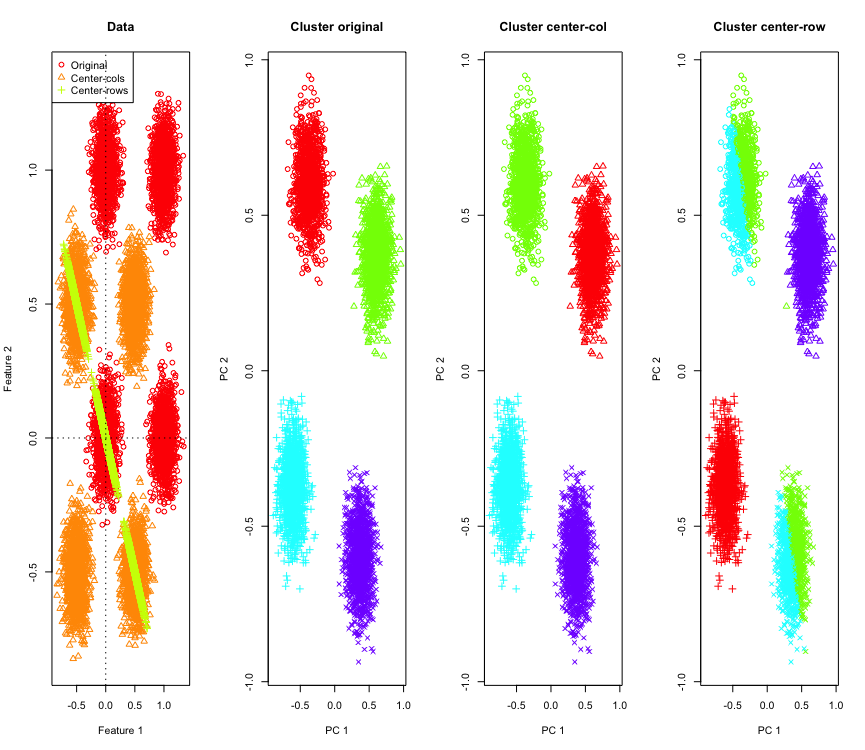
Para ilustrarlo, utilizaré datos gaussianos simulados en las esquinas de un hipercubo. Afortunadamente en R hay una función conveniente para eso (el código está al final de la respuesta). En el caso 2D es directo que los datos centrados en la media de las filas caerán en una línea que pasa por el origen a 135 grados. Los datos simulados son luego agrupados usando k-means con el número correcto de grupos. Los datos y los resultados del agrupamiento (visualizados en 2D utilizando PCA en los datos originales) se ven así (los ejes para el gráfico más a la izquierda son diferentes). Las formas diferentes de los puntos en los gráficos de agrupamiento se refieren a la asignación de grupos verdadera y los colores son el resultado del agrupamiento k-means.
![entrar descripción de la imagen aquí]()
Los grupos de la parte superior izquierda y la parte inferior derecha se dividen por la mitad cuando los datos se centran en la media de las filas. Por lo tanto, las distancias después de centrar en la media de las filas se distorsionan y no son muy significativas (al menos según el conocimiento de los datos).
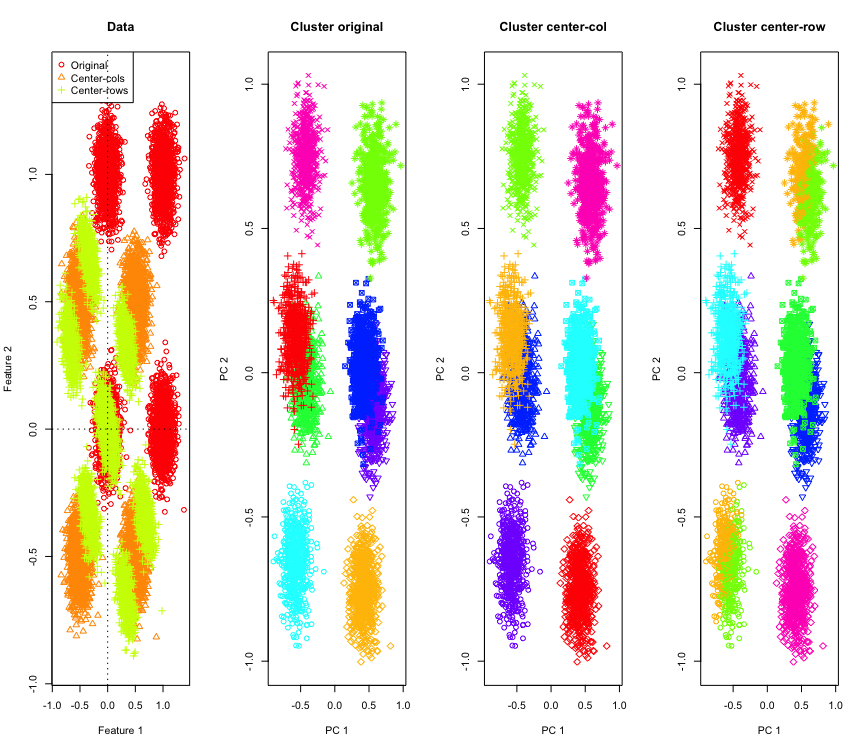
No es tan sorprendente en 2D, ¿qué sucede si usamos más dimensiones? Así es como se ve con datos 3D. La solución de agrupamiento después de centrar en la media de las filas es "mala".
![entrar descripción de la imagen aquí]()
Y similar con datos 4D (ahora no se muestra por brevedad).
¿Por qué está sucediendo esto? Centrar en la media de las filas empuja los datos hacia un espacio donde algunas características se acercan más de lo que lo hacen de otra manera. Esto debería reflejarse en la correlación entre las características. Veamos eso (primero en los datos originales y luego en los datos centrados en la media de las filas para los casos 2D y 3D).
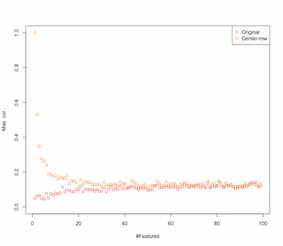
[,1] [,2] [1,] 1.000 -0.001 [2,] -0.001 1.000 [,1] [,2] [1,] 1 -1 [2,] -1 1 [,1] [,2] [,3] [1,] 1.000 -0.001 0.002 [2,] -0.001 1.000 0.003 [3,] 0.002 0.003 1.000 [,1] [,2] [,3] [1,] 1.000 -0.504 -0.501 [2,] -0.504 1.000 -0.495 [3,] -0.501 -0.495 1.000 Entonces parece que centrar en la media de las filas está introduciendo correlaciones entre las características. ¿Cómo se ve afectado esto por el número de características? Podemos hacer una simulación simple para averiguarlo. El resultado de la simulación se muestra a continuación (nuevamente el código al final).
![entrar descripción de la imagen aquí]()
Entonces, a medida que aumenta el número de características, el efecto de centrar en la media de las filas parece disminuir, al menos en cuanto a las correlaciones introducidas. Pero simplemente usamos datos aleatorios uniformemente distribuidos para esta simulación (como es común cuando se estudia la maldición de la dimensionalidad).
¿Qué sucede cuando usamos datos reales? Como muchas veces la dimensionalidad intrínseca de los datos es menor la maldición podría no aplicarse. En tal caso, supondría que centrar en la media de las filas podría ser una elección "mala", como se muestra arriba. Por supuesto, se necesita un análisis más riguroso para hacer afirmaciones definitivas.
Código para la simulación de agrupamiento
palette(rainbow(10))
set.seed(1024)
require(mlbench)
N <- 5000
for(D in 2:4) {
X <- mlbench.hypercube(N, d=D)
sh <- as.numeric(X$classes)
K <- length(unique(sh))
X <- X$x
Xc <- sweep(X,2,apply(X,2,mean),"-")
Xr <- sweep(X,1,apply(X,1,mean),"-")
show(round(cor(X),3))
show(round(cor(Xr),3))
par(mfrow=c(1,1))
k <- kmeans(X,K,iter.max = 1000, nstart = 10)
kc <- kmeans(Xc,K,iter.max = 1000, nstart = 10)
kr <- kmeans(Xr,K,iter.max = 1000, nstart = 10)
pc <- prcomp(X)
par(mfrow=c(1,4))
lim <- c(min(min(X),min(Xr),min(Xc)), max(max(X),max(Xr),max(Xc)))
plot(X[,1], X[,2], xlim=lim, ylim=lim, xlab="Característica 1", ylab="Característica 2",main="Datos",col=1,pch=1)
points(Xc[,1], Xc[,2], col=2,pch=2)
points(Xr[,1], Xr[,2], col=3,pch=3)
legend("topleft",legend=c("Original","Centro-columnas","Centro-filas"),col=c(1,2,3),pch=c(1,2,3))
abline(h=0,v=0,lty=3)
plot(pc$x[,1], pc$x[,2], col=rainbow(K)[k$cluster], xlab="PC 1", ylab="PC 2", main="Agrupamiento original", pch=sh)
plot(pc$x[,1], pc$x[,2], col=rainbow(K)[kc$cluster], xlab="PC 1", ylab="PC 2", main="Agrupamiento centro-columna", pch=sh)
plot(pc$x[,1], pc$x[,2], col=rainbow(K)[kr$cluster], xlab="PC 1", ylab="PC 2", main="Agrupamiento centro-fila", pch=sh)
}
Código para simulación de aumento de características
set.seed(2048)
N <- 1000
Cmax <- c()
Crmax <- c()
for(D in 2:100) {
X <- matrix(runif(N*D), nrow=N)
C <- abs(cor(X))
diag(C) <- NA
Cmax <- c(Cmax, max(C, na.rm=TRUE))
Xr <- sweep(X,1,apply(X,1,mean),"-")
Cr <- abs(cor(Xr))
diag(Cr) <- NA
Crmax <- c(Crmax, max(Cr, na.rm=TRUE))
}
par(mfrow=c(1,1))
plot(Cmax, ylim=c(0,1), ylab="Cor. máxima", xlab="#Características",col=1,pch=1)
points(Crmax, ylim=c(0,1), col=2, pch=2)
legend("topright", legend=c("Original","Centro-fila"),pch=1:2,col=1:2)
EDICIÓN
Después de buscar en Google, llegué a esta página donde las simulaciones muestran un comportamiento similar y propone que la correlación introducida por el centrado en la media de las filas sea $-1/(p-1)$.



1 votos
¿Puedes por favor citar la literatura que normaliza las filas? Me doy cuenta de que esta es una discusión relativamente antigua pero recientemente me encontré con un problema similar y estoy tratando de averiguar las diferencias. Publicaré mi opinión al respecto como una respuesta.
0 votos
Una mención de la normalización de filas se puede encontrar en p504 de Hastie, Tibshirani, Friedman en el contexto de agrupamiento: si las filas $\boldsymbol{x}$ y $\boldsymbol{x}'$ están centradas y normalizadas, la disimilitud $\ell^2$ produce el mismo orden de cercanía de puntos de datos que la similitud del coseno (= correlación por filas). De hecho, $\|\boldsymbol{x}\|=\|\boldsymbol{x}'\|=1$ implica que $\|\boldsymbol{x}-\boldsymbol{x}'\|^{2}=\|\boldsymbol{x}\|^{2}+\|\boldsymbol{x}'\|^{2}-2\langle\boldsymbol{x},\boldsymbol{x}'\rangle=2-2\cos\angle\boldsymbol{x}\boldsymbol{x}'$.