Me han llevado a creer (ver aquí y aquí) que la distancia de Mahalanobis es igual a la distancia euclidiana en los datos rotados por PCA. En otras palabras, tomando datos normales multivariados X, la distancia de Mahalanobis de todos los x desde cualquier punto dado (digamos 0) debería ser la misma que la distancia euclidiana de las entradas de Xrot desde 0, donde Xrot es el producto de los datos y la matriz de rotación de PCA.
1. ¿Es esto cierto?
Mi código a continuación me está sugiriendo que no lo es. En particular, parece que la varianza de la distancia de Mahalanobis alrededor de la distancia PCA-Euclidiana está aumentando en la magnitud de la distancia PCA-Euclidiana. ¿Es esto un error de codificación, o una característica del universo? ¿Tiene algo que ver con la imprecisión en una estimación de algo? ¿Algo que se eleva al cuadrado?
N=1000
cr = runif(1, min=-1, max=1)
A = matrix(c(1,cr,cr,1),2)
e<-mvrnorm(n = N, rep(0,2), A)
mx = apply(e, 2, mean)
sx = apply(e, 2, sd)
e = t(apply(e, 1, function(X){(X-mx)/sx}))
plot(e[,1], e[,2])
dum<-rep(0,2)
md = mahalanobis(e, dum, cov(e))
pc = prcomp(e, center=F, scale=F)
d<-as.matrix(dist(rbind(dum, pc$x), method='euclidean', diag=F))
d<-d[1,2:ncol(d)]
plot(d, md^.5)
abline(0,1)2. Si la respuesta a lo anterior es cierta, ¿se puede usar la distancia euclidiana PCA-rotada como un sustituto de la distancia de Mahalanobis cuando p>n?
Si no, ¿existe una métrica similar que capture la distancia multivariada, escalada por correlación, y para la cual existan resultados de distribución que permitan el cálculo de la probabilidad de una observación?
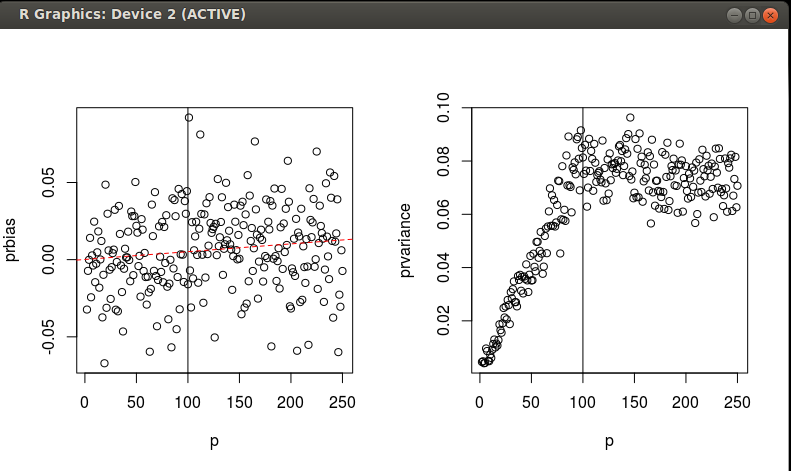
EDICIÓN He ejecutado algunas simulaciones para investigar la equivalencia de MD y SED en datos escalados/rotados a lo largo de un gradiente de n y p. Como mencioné anteriormente, estoy interesado en la probabilidad de una observación. Espero encontrar una buena forma de obtener la probabilidad de una observación que sea parte de una distribución normal multivariada, pero para la cual tengo datos nligeramentesesgadodelaMD,conunabuenacantidaddevarianzaqueparecedejardeaumentarcuandop=N$.
f = function(N=1000,n,p){
a = runif(p^2,-1,1)
a = matrix(a,p)
S = t(a)%*%a
x = mvrnorm(N, rep(0,p), S)
mx = apply(x, 2, mean)
sx = apply(x, 2, sd)
x = t(apply(x, 1, function(X){(X-mx)/sx}))
Ss = solve(cov(x))
x = x[sample(1:N, n, replace=F),]
md = mahalanobis(x, rep(0, p), Ss, inverted=T)
prMD<-pchisq(md, df = p)
pc = prcomp(x, center=F, scale=F)
d<-mahalanobis(scale(pc$x), rep(0, ncol(pc$x)), diag(rep(1, ncol(pc$x))))
prPCA<-pchisq(d, df = min(p, n))#N is the number of PCs where N`
Dos preguntas: 1. ¿Algún comentario sobre lo que estoy encontrando en estas simulaciones? 2. ¿Alguien puede formalizar lo que estoy encontrando con una expresión analítica para el sesgo y la varianza en función de n y p? Aceptaría una respuesta que haga esto.
`



4 votos
A partir de la fórmula misma de la distancia de Mahalanobis se sigue que es igual a la distancia euclidiana cuando la matriz de covarianza es la matriz identidad (o, para extender sin pérdida de clave - proporcional a la identidad). A menos que los datos sean exactamente esféricos, las covarianzas entre sus componentes principales son una matriz diagonal, no de identidad.
1 votos
Para tener en cuenta, el PCA no solo rota los datos, sino que también los escala de manera diferente en direcciones diferentes. La escala tiene un efecto en las mediciones de distancia.
2 votos
Las distancias cuadradas de Mahalanobis entre los puntos de datos son exactamente proporcionales a las distancias euclídeas cuadradas ponderadas calculadas en los componentes principales de los datos. El peso es
1/valor propiodel componente. (Y lo mismo ocurre también cuando hablamos de distancias entre puntos y centróide, en lugar de distancias punto a punto) Esta ponderación es lo que compensa las diferencias entre Mahalanobis y Euclidiana sobre las que he comentado.0 votos
@ttnphns Esa podría ser una respuesta para la primera parte de mi pregunta. Todavía estoy pensando en la segunda parte, sin embargo, si es un buen sustituto para mahalanobis cuando P>N. Estoy pensando, sin embargo, que la maldición de la dimensionalidad podría empezar a ser bastante severa.
1 votos
Puedes buscar en este sitio
Mahalanobis singularque podría proporcionar respuestas a tu segunda pregunta0 votos
¡Hola, @generic_user! Me pregunto si has tenido la oportunidad de ver mi respuesta a continuación. No dudes en preguntar si algo no queda claro.
0 votos
Hola Amoeba, vi y voté positivamente tu respuesta. Básicamente estás repitiendo lo que ttnphns dijo, cuando me recordó la escalación por los valores propios. Pero todavía estoy buscando una buena forma de pensar en cómo interpretar la distancia euclidiana en el espacio transformado cuando $n
0 votos
He actualizado mi respuesta a tu Q2, por favor échale un vistazo. Si esto no te satisface, te animo a editar tu pregunta para aclarar tu Q2, tal vez puedas intentar explicar qué te preocupa aquí y qué sigue sin estar claro.
0 votos
Edición interesante. Si tu código produce alguna(s) figura(s), ¿podrías quizás insertarlas en tu pregunta? No todos son usuarios de R...
0 votos
@generic_user. Gracias. ¿Puede explicar por favor cuál es el significado de la distancia de Mahalanobis en el espacio de componentes principales? ¿Es diferente de la distancia de Mahalanobis en el espacio de datos original? ¿Qué ventajas tendríamos al tener la distancia de Mahalanobis en cualquiera de los espacios?