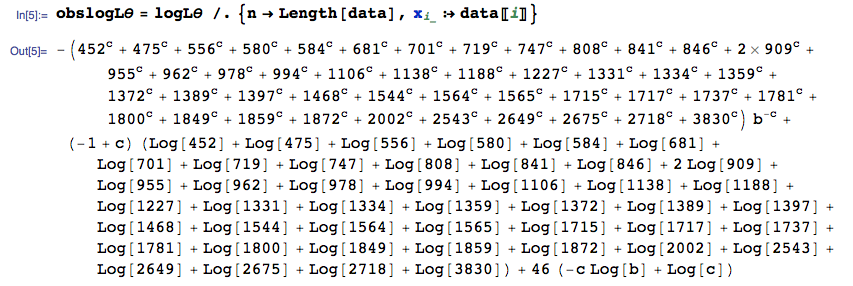
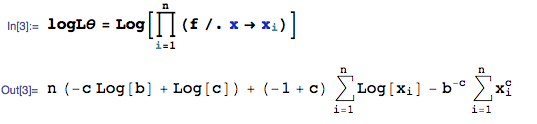Tengo los siguientes datos Y que quiero conseguir un MLE estimación de los parámetros a través de una distribución de Weibull en R.
1468, 1872, 475, 1372, 3830, 1849, 978, 1389, 909, 701, 1227, 962, 1781, 580, 584, 2675, 841, 1544, 452, 955, 556, 1737, 747, 1565, 1331, 1188, 2649, 1800, 2718, 808, 1138, 909, 1359, 846, 1334, 1397, 719, 1715, 681, 2002, 994, 2543, 1564, 1717, 1106, 1859
Si intento ejecutar fitdistr(Y, "weibull") me sale un aviso:
fit = fitdistr(Y, "weibull")
Warning message:
In densfun(x, parm[1], parm[2], ...) : NaNs produced
> warnings(fit)
Warning message:
In densfun(x, parm[1], parm[2], ...) : NaNs produced Error in
at(list(...), file, sep, fill, labels, append) :
argument 2 (type 'list') cannot be handled by 'cat'
Pero todavía me da un MLE. Sin embargo, el valor es diferente del resultado SAS da.
la salida de R:
shape scale
2.1103684 1537.2344072
(0.2245888) (112.1596367)
salida de SAS (usando proc lifereg):
Weibull Scale 1550.559
Weibull Shape 2.1195
Cuál es la causa de la discrepancia y son los preferidos de los paquetes/funciones para calcular estimaciones sencillas de Emv para las distribuciones más MASS y fitdistr?