Estoy ejecutando un modelo mixto lineal generalizado con familia beta sobre el efecto de la cubierta superior (proporción (0,1)) en la proporción de aves que carroñan de carrona dejada en la naturaleza (proporción (0,1)), con Área como factor aleatorio (factor w/ 6 niveles).
> myglmm <- glmmTMB(ProportionBirdsScavenging ~ OverheadCover + (1|Area), data = df_prop_birds_eating, beta_family(link = "logit"), weights = pointWeight_scaled)
> summary(myglmm)
Family: beta ( logit )
Formula: ProportionBirdsScavenging ~ OverheadCover + (1 | Area)
Data: df_prop_birds_eating
Weights: pointWeight_scaled
AIC BIC logLik deviance df.resid
-5.3 0.8 6.7 -13.3 30
Random effects:
Conditional model:
Groups Name Variance Std.Dev.
Area (Intercept) 1.198e-10 1.094e-05
Número de observaciones: 34, grupos: Area, 6
Parámetro de sobredispersión para la familia beta (): 5.17
Modelo condicional:
Estimado Error Estándar valor z valor p
(Intercepto) 1.7869 0.7196 2.483 0.013017 *
OverheadCover -4.7387 1.2661 -3.743 0.000182 ***
---
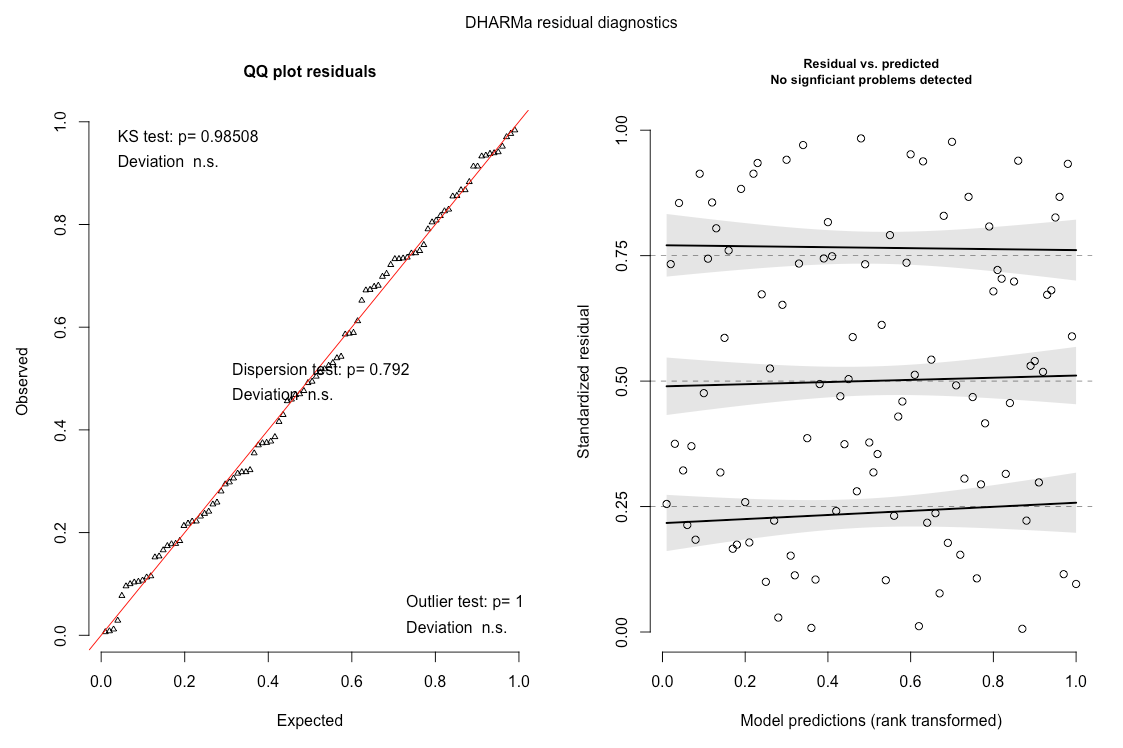
Códigos de significancia: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1El valor del parámetro de sobredispersión es 5.17. He intentado encontrar información sobre este parámetro para modelos beta, pero no pude encontrar mucho. La mayoría de lo que encontré fue sobre la distribución poisson o binomial, y pruebas sobre la significancia ej. DHARMa::testOverdispersion, performance::check_overdispersion, y AER::dispersiontest solo prueban para GLMs de Poisson Mi pregunta es si este valor de parámetro de sobredispersión de 5.17 es demasiado alto? ¿Significa esto que las suposiciones del modelo no se cumplen y la salida no se puede confiar? En caso afirmativo, ¿hay alguna forma de solucionarlo para que mi modelo produzca resultados confiables?
Sobre la sobredispersión en modelos de Poisson, con frecuencia leo que agregar un parámetro de dispersión 'arreglaría' la sobredispersión, pero en el modelo beta que estoy utilizando ya hay un parámetro de dispersión. ¿Alguien puede explicar esto?