Puedes visualizar el árbol de decisión con Graphviz
![]()
que sklearn muestra cómo hacer así:
from sklearn.datasets import load_iris
from sklearn import tree
iris = load_iris()
X, y = iris.data, iris.target
clf = tree.DecisionTreeClassifier()
clf = clf.fit(X, y)
dot_data = tree.export_graphviz(clf, out_file=None,
feature_names=iris.feature_names,
class_names=iris.target_names,
filled=True, rounded=True,
special_characters=True)
graph = graphviz.Source(dot_data)
graph.view()
Cada nodo contiene la siguiente información:
- regla de decisión (por ejemplo petal length (cm)≤2.45petal length (cm)≤2.45 )
- Impureza de Gini, ∑ipi(1−pi)∑ipi(1−pi)
- número de muestras que llegan al nodo (p. ej.
samples=54 )
- recuento de miembros de cada clase (por ejemplo
value = [0,2,4] )
- clase dominante (o al menos no dominada) (p. ej.
class=versicolor )
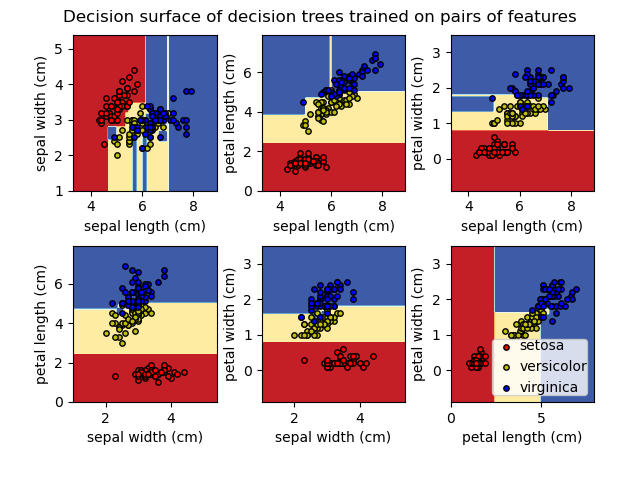
Y también puedes visualizar el límite de decisión
![]()
que sklearn nos muestra que se puede hacer así:
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from sklearn.tree import DecisionTreeClassifier
from sklearn.inspection import DecisionBoundaryDisplay
iris = load_iris()
# Parameters
n_classes = 3
plot_colors = "ryb"
plot_step = 0.02
for pairidx, pair in enumerate([[0, 1], [0, 2], [0, 3], [1, 2], [1, 3], [2, 3]]):
# We only take the two corresponding features
X = iris.data[:, pair]
y = iris.target
# Train
clf = DecisionTreeClassifier().fit(X, y)
# Plot the decision boundary
ax = plt.subplot(2, 3, pairidx + 1)
plt.tight_layout(h_pad=0.5, w_pad=0.5, pad=2.5)
DecisionBoundaryDisplay.from_estimator(
clf,
X,
cmap=plt.cm.RdYlBu,
response_method="predict",
ax=ax,
xlabel=iris.feature_names[pair[0]],
ylabel=iris.feature_names[pair[1]],
)
# Plot the training points
for i, color in zip(range(n_classes), plot_colors):
idx = np.where(y == i)
plt.scatter(
X[idx, 0],
X[idx, 1],
c=color,
label=iris.target_names[i],
cmap=plt.cm.RdYlBu,
edgecolor="black",
s=15,
)
plt.suptitle("Decision surface of decision trees trained on pairs of features")
plt.legend(loc="lower right", borderpad=0, handletextpad=0)
_ = plt.axis("tight")
La documentación de sklearn utiliza proyecciones ortogonales estándar. En principio, puede buscar otras proyecciones que pueden implicar primero la rotación u otras operaciones.
La primera visualización puede dar una idea procedimental de lo que hace un árbol de decisión, y la segunda puede dar una idea espacial de cómo el modelo ha dividido el espacio.