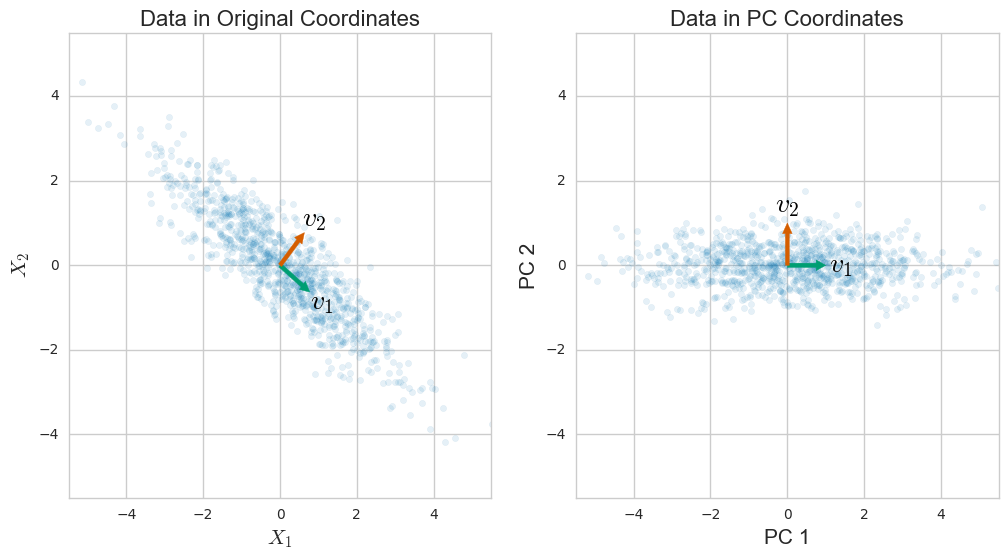
Permítanme comenzar con el PCA. Supongamos que tenemos n puntos de datos compuestos por d números (o dimensiones) cada uno. Si se centran estos datos (se resta el punto de datos medio $\mu$ de cada vector de datos $x_i$ ) puedes apilar los datos para hacer una matriz
$$ X = \left( \begin{array}{ccccc} && x_1^T - \mu^T && \\ \hline && x_2^T - \mu^T && \\ \hline && \vdots && \\ \hline && x_n^T - \mu^T && \end{array} \right)\,. $$
La matriz de covarianza
$$ S = \frac{1}{n-1} \sum_{i=1}^n (x_i-\mu)(x_i-\mu)^T = \frac{1}{n-1} X^T X $$
mide hasta qué punto las diferentes coordenadas en las que se dan los datos varían conjuntamente. Así que quizá no sea sorprendente que el ACP, que está diseñado para captar la variación de los datos, pueda darse en términos de la matriz de covarianza. En particular, la descomposición de valores propios de $S$ resulta ser
$$ S = V \Lambda V^T = \sum_{i = 1}^r \lambda_i v_i v_i^T \,, $$
donde $v_i$ es el $i$ -a Componente principal o PC, y $\lambda_i$ es el $i$ -valor propio de $S$ y también es igual a la varianza de los datos a lo largo del $i$ -Cuarto PC. Esta descomposición proviene de un teorema general del álgebra lineal, y de algunos trabajos hace que hay que hacer para motivar al relatino a la PCA.
![PCA of a Randomly Generated Gaussian Dataset]()
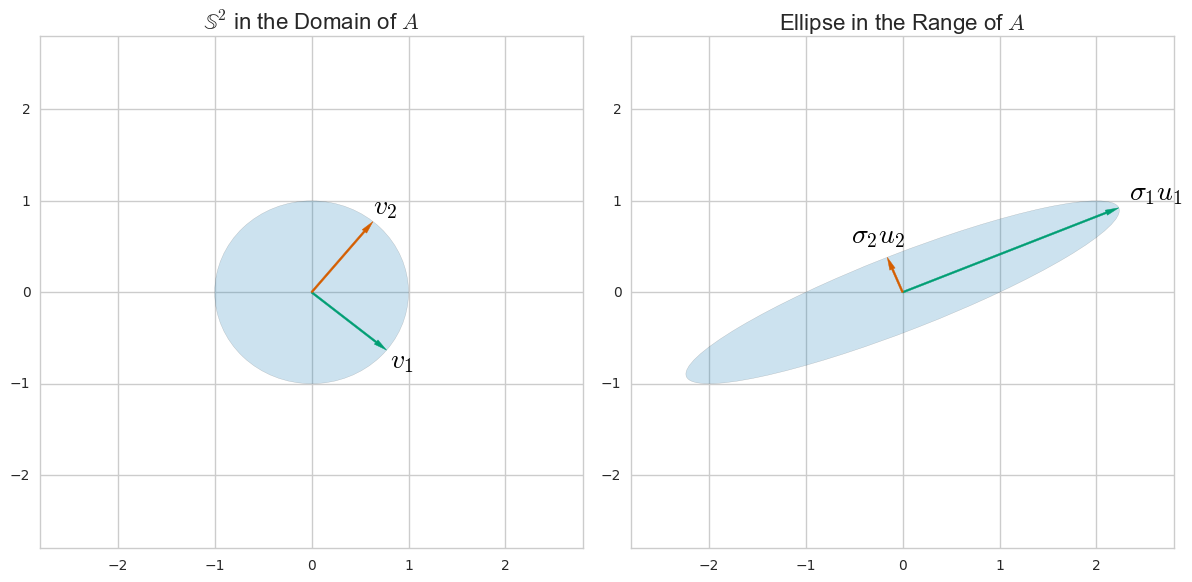
La SVD es una forma general de entender una matriz en términos de su espacio de columnas y espacio de filas. (Es una forma de reescribir cualquier matriz en términos de otras matrices con una relación intuitiva con el espacio de filas y columnas). Por ejemplo, para la matriz $A = \left( \begin{array}{cc}1&2\\0&1\end{array} \right)$ podemos encontrar direcciones $u_i$ y $v_i$ en el dominio y el rango para que
![SVD for a 2x2 example]()
Puede encontrarlos teniendo en cuenta cómo $A$ como una transformación lineal transforma una esfera unitaria $\mathbb S$ en su dominio a una elipse: los semiejes principales de la elipse se alinean con el $u_i$ y el $v_i$ son sus preimágenes.
En cualquier caso, para la matriz de datos $X$ arriba (en realidad, sólo hay que poner $A = X$ ), la SVD nos permite escribir
$$ X = \sum_{i=1}^r \sigma_i u_i v_j^T\,, $$
donde $\{ u_i \}$ y $\{ v_i \}$ son conjuntos ortonormales de vectores.Una comparación con la descomposición de valores propios de $S$ revela que los "vectores singulares derechos" $v_i$ son iguales a los PC, los "vectores singulares derechos" son
$$ u_i = \frac{1}{\sqrt{(n-1)\lambda_i}} Xv_i\,, $$
y los "valores singulares" $\sigma_i$ se relacionan con la matriz de datos a través de
$$ \sigma_i^2 = (n-1) \lambda_i\,. $$
Es un hecho general que los vectores singulares de la derecha $u_i$ abarcan el espacio de las columnas de $X$ . En este caso concreto, $u_i$ nos dan una proyección a escala de los datos $X$ en la dirección del $i$ -ésima componente principal. Los vectores singulares de la izquierda $v_i$ en general abarcan el espacio de filas de $X$ , lo que nos da un conjunto de vectores ortonormales que abarcan los datos de forma parecida a los PC.
Me refiero a algunos detalles más y a los beneficios de la relación entre PCA y SVD en este artículo más largo .




14 votos
Escribí esta pregunta tipo FAQ junto con mi propia respuesta, porque se está preguntando con frecuencia en varias formas, pero no hay un hilo canónico y así cerrar los duplicados es difícil. Por favor, proporcione meta comentarios en este hilo meta que lo acompaña .
3 votos
stats.stackexchange.com/questions/177102/
2 votos
Además de una excelente y detallada respuesta de la ameba con sus enlaces adicionales podría recomendar para comprobar este donde el PCA se considera junto a otras técnicas basadas en el SVD. La discusión allí presenta un álgebra casi idéntica a la de ameba con la única diferencia de que el discurso allí, al describir PCA, va sobre la descomposición SVD de $\mathbf X/\sqrt{n}$ [o $\mathbf X/\sqrt{n-1}$ ] en lugar de $\bf X$ - que es simplemente conveniente ya que se relaciona con el PCA realizado a través de la eigendecomposición de la matriz de covarianza.
0 votos
El PCA es un caso especial del SVD. El PCA necesita los datos normalizados, idealmente la misma unidad. La matriz es nxn en PCA.
1 votos
@OrvarKorvar: ¿De qué matriz n x n estás hablando?
0 votos
@Cbhihe el
n x nes la matriz de covarianza de la matriz de datosX