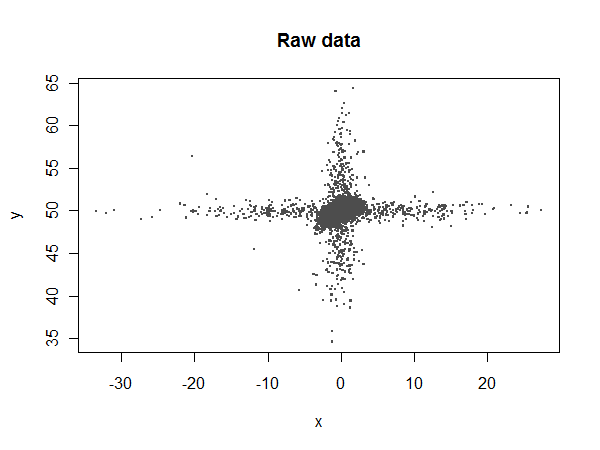
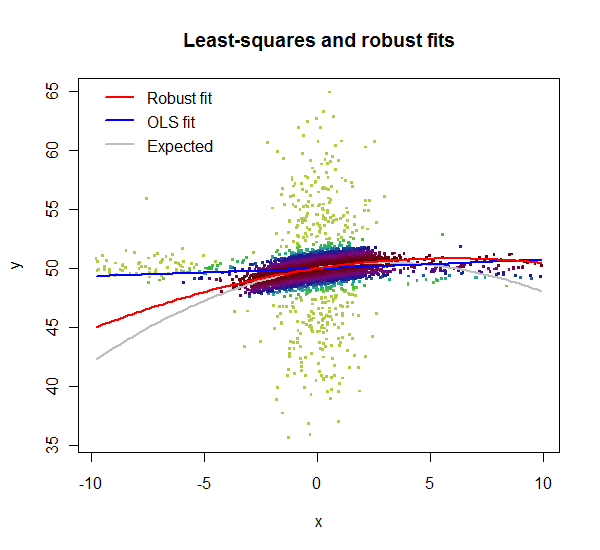
Tengo un conjunto de datos para Temperatura y KwH y actualmente estoy realizando la regresión a continuación. (se realiza una regresión adicional basada en los coeficientes dentro de PHP)
# Alguna especie de estructura de lista..
UsageDataFrame <- data.frame(Energía, Temperaturas);
# lm se usa para ajustar modelos lineales. Se puede utilizar para llevar a cabo regresiones,
# análisis de varianza de una sola estratificación y análisis de covarianza (aunque
# aov puede proporcionar una interfaz más conveniente para estos).
LinearModel <- lm(Energía ~ 1+Temperaturas+I(Temperaturas^2), data = UsageDataFrame)
# coeficientes
Coeficientes <- coeficientes(LinearModel)
system('clear');
cat("--- Coeficientes ---\n");
print(Coeficientes);
cat('\n\n');El problema viene con nuestros datos, no podemos asegurar que no haya fallas de comunicación aleatorias o simplemente errores aleatorios. Esto puede dejarnos con valores como
Temperaturas <- c(16,15,13,18,20,17,20);
Energía <- c(4,3,3,4,0,60,4)
Temperaturas <- c(17,17,14,17,21,16,19);
Energía <- c(4,3,3,4,0,0,4)Ahora, como humanos, podemos ver claramente que el 60 para Kwh es un error basado en la temperatura, sin embargo tenemos más de 2,000 sistemas cada uno con múltiples medidores y cada uno en diferentes lugares en todo el país... y con diferentes niveles de uso normal de energía.
Un conjunto de datos normal tendría 48 valores tanto para Temperaturas como para Energía por día, por medidor. En un año completo, es probable que podamos tener alrededor de 0-500 puntos malos por medidor de un total de 17520 puntos.
He leído otros posts sobre el paquete tawny sin embargo no he visto realmente ejemplos que me permitan pasar un data.frame y procesarlos a través de un análisis cruzado.
Entiendo que no se puede hacer mucho, sin embargo seguramente se podrían eliminar los valores muy grandes en función de la temperatura? Y la cantidad de veces que ocurre..
Dado que R se basa en matemáticas no veo ninguna razón para trasladarlo a otro lenguaje.
Tenga en cuenta: Soy un desarrollador de software y nunca he usado R antes.
-- Editar --
Bien, aquí hay un ejemplo del mundo real, parece que este medidor es un buen ejemplo. Se puede ver que los ceros se están acumulando y luego se inserta un valor masivo. "23, 65, 22, 24" son ejemplos de esto. Esto sucede cuando hay una falla de comunicación y el dispositivo retiene el valor de los datos y continúa sumándolos.
(Solo para decir que las fallas de comunicación están fuera de mi alcance ni puedo cambiar el software)
Sin embargo, dado que el cero es un valor válido, quiero eliminar cualquier número masivo en contra de las temperaturas o ceros donde está claro que son un error.
La idea de detectar esto y promediar los datos de nuevo no es una solución para esto tampoco, sin embargo se discutió, pero dado que los datos de este medidor son cada 30 minutos y las fallas de comunicación pueden ocurrir durante días.
La mayoría de los sistemas están usando más Energía que esto, así que tal vez sea un mal ejemplo desde el punto de vista de eliminar los ceros.
Energía: http://pastebin.com/gBa8y5sM Temperaturas: http://pastie.org/4371735
(Pastebin parece haber dejado de funcionar para mí después de publicar un archivo tan grande)